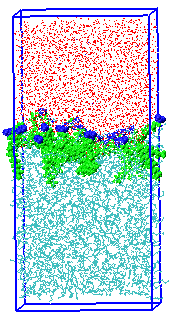
体系:油水+表活剂体系,如下图:

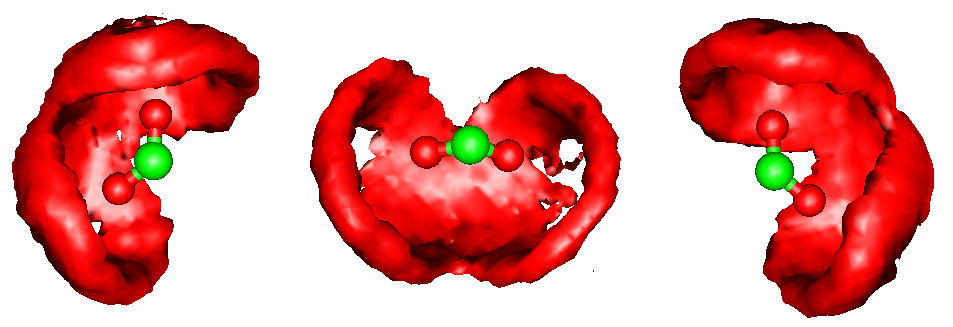
目标:计算表活剂头基C原子附近水分子的SDF,类似于下图:
计算步骤:
使用gmx make_ndx创建两个组, 一个包含中心分子, 一个包含要统计SDF的原子
我试过两种方案,一种是以一个表活剂作为中心分子建一个group,以中心分子头基附近的水建一个group,用这两个group计算SDF;另一种是以所有表活剂作为中心分子建一个group,以体系中所有水建一个group,用这两个group计算SDF。最后结果都差不多。
最终选中心分子为COO, SDF的原子为体系中所有的水
使中心分子在盒子内居中, 同时所有其他分子处于盒子内
gmx_mpi trjconv -s md0.tpr -f md0.trr -n sdf.ndx -o md0_cnt.xtc -center -ur compact -pbc mol
Select group for centering时选择中心分子组, Select group for output时选择System组
按中心分子对轨迹进行叠合, 移除中心分子的转动和平动
gmx_mpi trjconv -s md0.tpr -f md0_cnt.xtc -n sdf.ndx -o md0_cnt_fit.xtc -fit rot+trans
Select group for least squares fit时选择中心分子组, Select group for output时选择System组
统计分布
gmx_mpi spatial -s md0.tpr -f md0_cnt_fit.xtc -n sdf.ndx -nab 80 -b 15000 -e 18000
Select group to generate SDF时选择要统计SDF的组, Select group to output coords (e.g. solute)时选择中心分子组
注意: 使用gmx spatial计算空间分布函数时, 在模拟过程中要尽量多输出些轨迹(约5000帧,根据结果可自行调节输出的帧数), 这样得到的SDF才可能光滑.
grid.cube文件, 以等值面模式查看结果步骤来源: VMD画cube文件等密度面图(轨道或电子密度等)
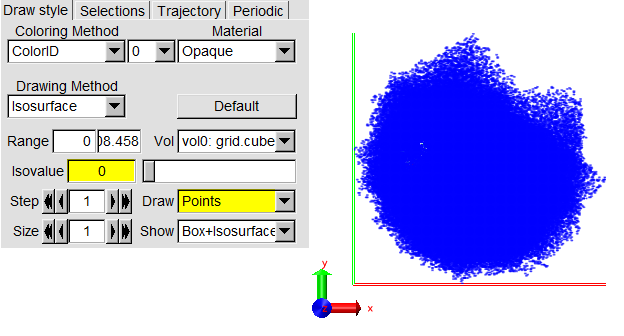
new molecule打开文件。Graphics下的representations.在弹出的对话框中,Drawing method选cpk,使画出的分子更漂亮。图(以所有表活剂作为中心分子建一个group,以体系中所有水建一个group的结果)如下:
点Create Rep,新建一个Representation.
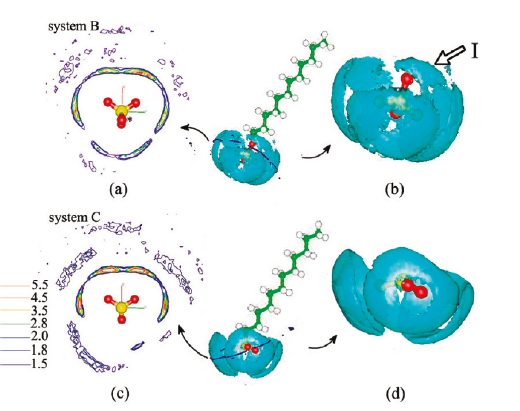
选新建的Representation后,Drawing method选isosurface. 先画正的部分,即在isovalue中输入一个正的值如0.02。在Coloring method中选择ColorID,为这个面指定颜色。图(表活剂包裹在里面)如下:
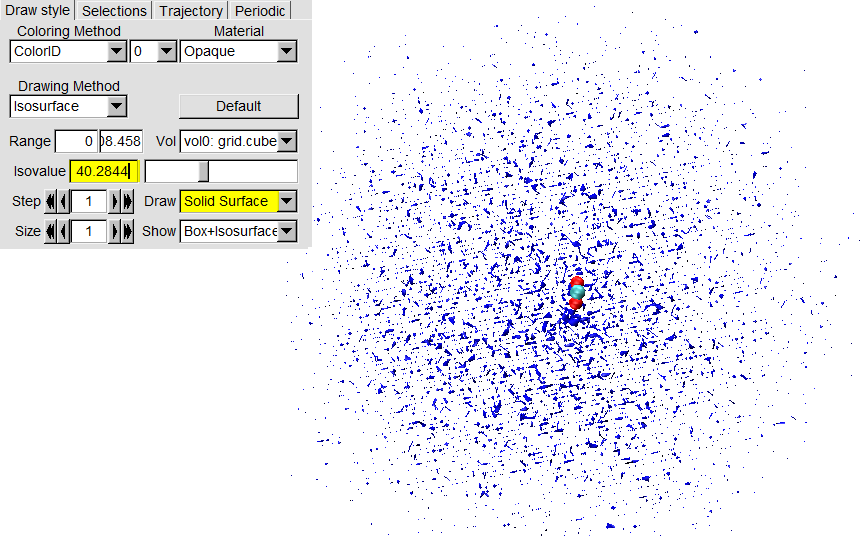
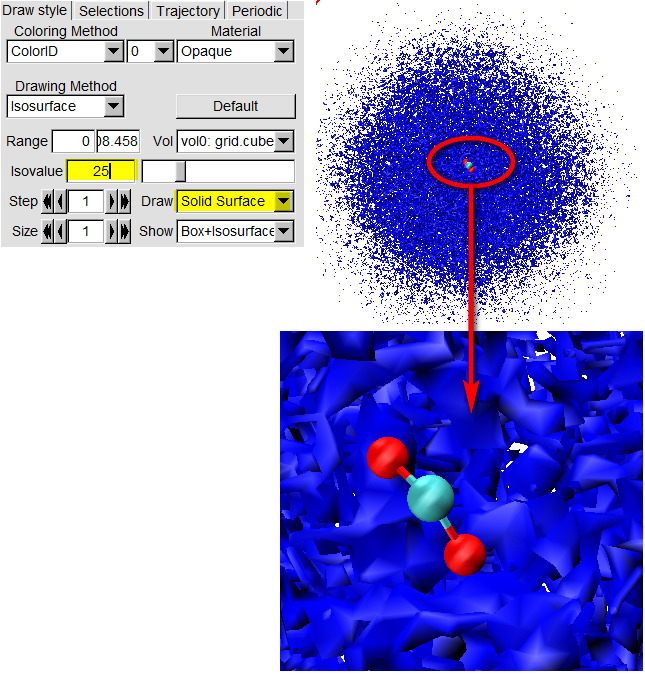
【李继存 注】等值面的具体数值要根据自己的计算结果确定, 选择一个有代表性且较光滑的等值面即可. 由于示例数据尚未收敛, 所以结果看起来并不好.
点Create Rep,再新建一个Representation.
选新建的Representation后,Drawing method选isosurface. 再画负的部分,即在isovalue中输入一个负值如-0.02。在Coloring method中选择ColorID,为这个面指定颜色。如下图所示:
【李继存 注】对SDF, 由于不会出负值, 所以此步骤可忽略.
在Graphics中选择Colors, 在Catagories中选displays,Name中选择backgroud,指定背景的颜色。也可在display菜单下的backgroud指定背景为梯度颜色。
render.将grid.cube文件转化成grid.plt文件
操作: 菜单栏run | {gCube2plt/g94cub2pl (cube)}, 浏览输入文件grid.cube, 指定输出文件, 命名为grid.plt. 然后点击最下面的Apply. 这样就转化成plt格式的文件了, 与此同时还生成了一个grid.crd文件, 这个crd文件作为结构文件.
读入上一步的grid.crd文件: File | Import | Coords
选择Plot|Contour, 然后import, 最后根据需要调节contour levels value及颜色, 并且可通过detail调节曲面的光滑度.
最终结果