
DISCLAIMER: Permission is hereby granted, free of charge, to any person in academic and/or industrial research and science (independent of their field of interest, country of origin, ethnic group or political system) to obtain a copy of this information and all associated documentation, files, models and data, to deal with the information without restriction (that is to say with no limits to the rights to use, copy, modify, merge, publish, distribute and sublicense copies of the information) and to permit persons to whom the information is furnished to do so, subject to the following conditions: The above copyright notice and this permission notice shall be included in all copies of substantial portions of the information. THE INFORMATION IS PROVIDED "AS IS", NOT PEER-REVIEWED, WITHOUT WARRANTY OF ANY KIND, EXPRESS OR IMPLIED, INCLUDING BUT NOT LIMITED TO THE WARRANTIES OF MERCHANTABILITY, FITNESS FOR A PARTICULAR PURPOSE AND NONINFRINGEMENT. IN NO EVENT SHALL THE AUTHORS OR COPYRIGHT HOLDERS BE LIABLE FOR ANY CLAIM, DAMAGES OR OTHER LIABILITY, WHETHER IN AN ACTION OF CONTRACT, TORT OR OTHERWISE, ARISING FROM, OUT OF OR IN CONNECTION WITH THE INFORMATION. PARTS OF BACKGROUND INFORMATION SECTION ARE DERIVED FROM PUBLIC SOURCES.
武汉冠状病毒(WHO 2019-nCoV)1于2019年首次报道, 是一种正链单股RNA冠状病毒, 该病毒爆发于中国武汉, 并由此向外传播. 截至2020年1月23日, 中国政府封锁了武汉市, 黄冈市和鄂州市(总人口约1500万)以遏制病毒爆发2, 3. 负责调查疫情的中国卫生委员会的负责人钟南山说: 广东已经确认了病毒可以在人与人之间传播4. 目前尚无针对新病毒的特定治疗方法, 但可能会尝试使用现有的抗病毒药物5.
据维基百科6, 武汉β-冠状病毒的序列与在蝙蝠中发现的β-冠状病毒相似. 但是, 该病毒在遗传上不同于其他冠状病毒, 如 严重急性呼吸综合征相关冠状病毒(SARS) 和 中东呼吸综合征相关冠状病毒(MERS). 与SARS冠状病毒类似, 武汉β-冠状病毒属于β冠状病毒B属(即Sarbecovirus亚属). 中国疾病预防控制中心, 病原生物学研究所和武汉金银潭医院已分离报道了18种新的冠状病毒基因组, 包括BetaCoV/Wuhan/IVDC-HB-01/2019, BetaCoV/Wuhan/IVDC-HB-04/2020, BetaCoV/Wuhan/IVDC-HB-05/2019, BetaCoV/Wuhan/WIV04/2019, BetaCoV/Wuhan/IPBCAMS-WH-01/2019, 其RNA序列长度约30 kb.
基于获得的新基因组, 已经对nCoV Spike(S)蛋白的受体结合蛋白(RBD)进行了多次蛋白质建模. 截至2020年1月23日, 中国的两个研究组认为S蛋白与SARS受体(血管紧张素转换酶2, ACE2)的亲和力足够大, 病毒因此得以进入细胞.
进入细胞后, 病毒会复制RNA基因组, 形成一个长的多聚蛋白, 所有的蛋白都附着于此. 冠状病毒具有一个非结构性蛋白(蛋白酶), 它能够分离链中的蛋白. 对于病毒, 这是基因上较为经济的一种做法, 能够以少量的核苷酸编码最大数量的基因7.
香港大学医学院教授此前曾表示, 早些时候已经对SARS进行了研究, 发现蛋白酶抑制剂和其他药物可以有效地治疗由SARS, MERS以及其他冠状病毒导致的呼吸系统疾病.
有6种冠状病毒可以感染人类, 还有24种其他类型的冠状病毒可以感染动物, 包括蝙蝠, 鸟类, 大鼠和牛. 他说, 由于大多数武汉患者与华南海鲜市场有联系, 这种未知的冠状病毒极有可能从蝙蝠传播给野生动物, 并在变异后传播给人类. 此外, 他表示, 通常情况下, 新的疾病在人与人之间的传染性不会很高, 所以只有与患者密切接触的人才会被感染. 如果武汉的疾病与SARS相似, 那么可以通过使用利巴韦林, 蛋白酶抑制剂和干扰素来治愈患者8.
哈佛大学公共卫生学院的流行病学家迈克尔·米纳(Michael Mina)昨天表示, 他了解到中国的某些患者正在接受蛋白酶抑制剂, 抗病毒药物的治疗, 这些药物都是为治疗HIV感染者而开发的, 曾用于治疗SARS且取得了一定的成功9.
在这种情况下, Innophore决定动用大量的人力和计算资源来支持建模工作. 尽管Innophore并不专注于全球流行病领域, 但我们的主要专长--结构酶学的基本原理本身与应用领域无关. 在过去的几年中, 我们有机会与化学, 制药, 营养和农业领域的众多学术和企业合作伙伴就涉及蛋白酶的许多不同酶类进行了合作研究. 我们致力于研究消费产品中使用的蛋白酶, 用于生物催化的蛋白酶以及人和猿类IV蛋白酶, 并研究相互作用的动力学, 尤其是HIV1蛋白酶10与经典抑制剂(如Mozenavir)的各种立体异构体之间的相互作用.
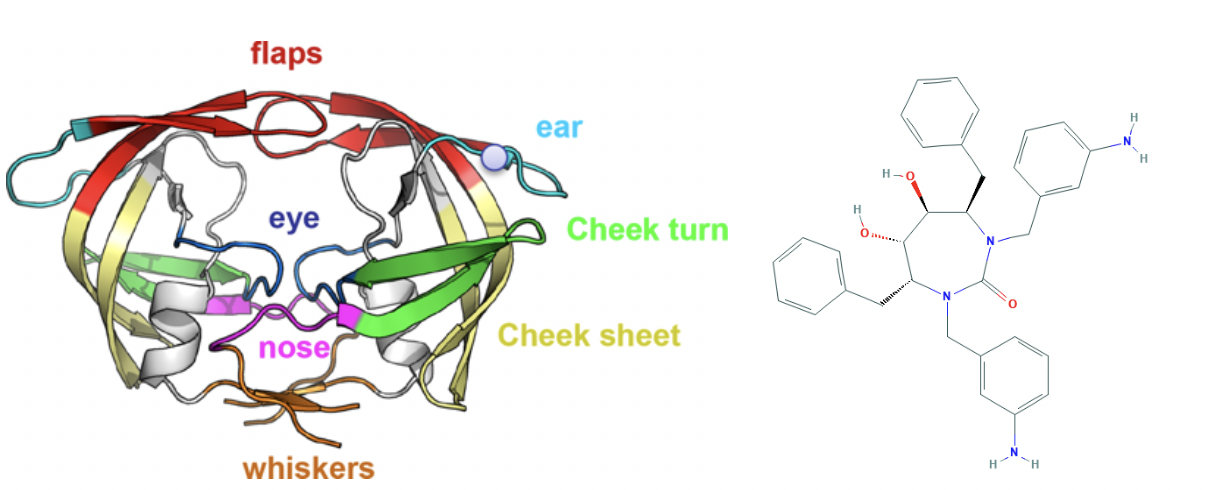
左: HIV-1蛋白酶的拓扑结构11, 右: HIV-1蛋白酶抑制剂DMP-450/Mozenavir12
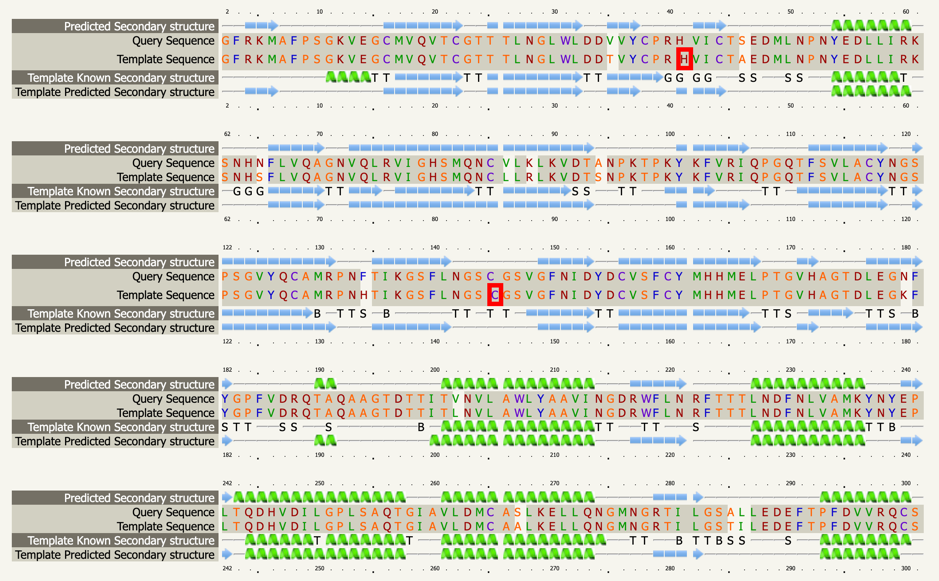
尽管已经有针对这种病毒的建模, 如13, 14, 我们仍决定重新从头开始以避免任何潜在的偏见, 并专注于自己团队最熟悉的蛋白质类别: 2019-nCoV的病毒蛋白酶. 普渡大学的癌症结构生物学教授, 生物化学系主任安德鲁·梅斯卡(Andrew Mesecar)也正在研究该蛋白酶的结构预测以及它与潜在抑制剂的相互作用. 我们正在等待这些结构公开发布. 与此同时, 我们分析了Wu, F.等人报道的来自武汉海鲜市场的肺炎病毒的基因组(NCBI基因组ID MN90894715, GenBank: MN908947.3, LOCUS MN908947, 29903 bp, ss-RNA linear VRL 23-JAN-2020), 基于与已知SARS冠状病毒蛋白酶的多重序列比对, 我们确定了潜在的蛋白酶序列. 下图给出了具有2019-nCoV orf1ab多聚蛋白和蛋白ID QHD43415.116(PDB结构5N5O17)的比对序列区域. 5N5O是由德国感染研究中心的Zhang, L.和Hilgenfeld R.在2017年使用Clustal O提取的一种SARS冠状病毒的主要蛋白酶结构:
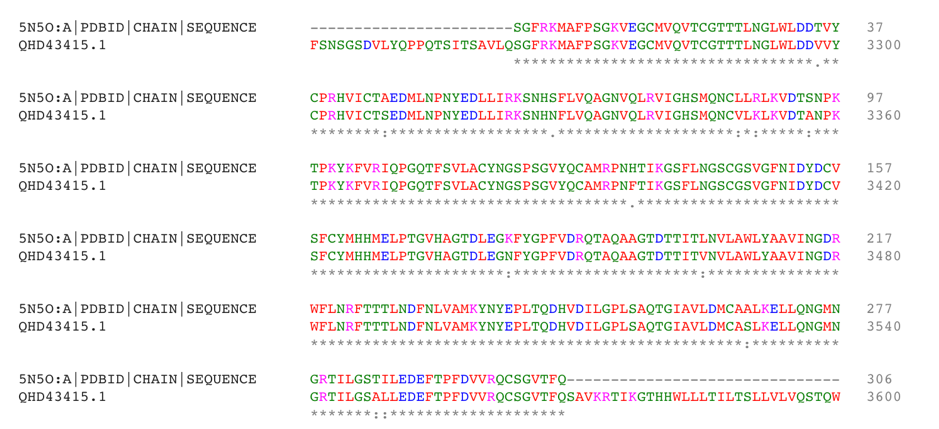
使用EMBOSS Needle将翻译后的2019-nCoV基因序列与另一个PDB结构3TLO18(HCoV-NL63 3C样蛋白酶的晶体结构)进行比对, 我们得到了相同的比对区域:
# Length: 7097 长度
# Identity: 136/7097 ( 1.9%) 识别
# Similarity: 192/7097 ( 2.7%) 相似度
# Gaps: 6795/7097 (95.7%) 间隙
# Score: 651.5 得分

从位置X到Y提取推定的蛋白酶序列得到了306个氨基酸的推定蛋白酶序列, 其分子量约33.8 kDa, 处于典型蛋白酶的上限.
>由5N5O推测的蛋白酶: 序列比对
>QHD43415.1 putative protease by 5N5O:A sequence alignment
SGFRKMAFPSGKVEGCMVQVTCGTTTLNGLWLDDVVYCPRHVICTSEDMLNPNYEDLLIRKSNHNFLVQ
AGNVQLRVIGHSMQNCVLKLKVDTANPKTPKYKFVRIQPGQTFSVLACYNGSPSGVYQCAMRPNFTIKG
SFLNGSCGSVGFNIDYDCVSFCYMHHMELPTGVHAGTDLEGNFYGPFVDRQTAQAAGTDTTITVNVLAW
LYAAVINGDRWFLNRFTTTLNDFNLVAMKYNYEPLTQDHVDILGPLSAQTGIAVLDMCASLKELLQNGM
NGRTILGSALLEDEFTPFDVVRQCSGVTFQ
再次将该序列与PDB进行比对, 发现了序列相似性非常高, 分辨率足够好的蛋白质, 可用于随后的同源建模, 例如PDB结构2A5K19.
PDB序列搜索
PDB Sequence Search:
SGFRKMAFPS GKVEGCMVQV TCGTTTLNGL WLDDVVYCPR HVICTSEDML NPNYEDLLIR KSNHNFLVQA GNVQLRVIGH SMQNCVLKLK VDTANPKTPK YKFVRIQPGQ TFSVLACYNG SPSGVYQCAM RPNFTIKGSF LNGSCGSVGF NIDYDCVSFC YMHHMELPTG VHAGTDLEGN FYGPFVDRQT AQAAGTDTTI TVNVLAWLYA AVINGDRWFL NRFTTTLNDF NLVAMKYNYE PLTQDHVDIL GPLSAQTGIA VLDMCASLKE LLQNGMNGRT ILGSALLEDE FTPFDVVRQC SGVTFQ
Expectation Value = 10.0, Sequence Identity = 0%, Search Tool = blast, Mask Low Complexity=yes)

总共发现了138个结构满足一般期望值10:
1LVO, 1P9S, 1P9U, 1Q2W, 1UJ1, 1UK2, 1UK3, 1UK4, 1WOF, 1Z1I, 1Z1J, 2A5A, 2A5I, 2A5K, 2ALV, 2AMD, 2AMP, 2AMQ,
2BX3, 2BX4, 2C3S, 2D2D, 2DUC, 2GT7, 2GT8, 2GTB, 2GX4, 2GZ7, 2GZ8, 2GZ9, 2H2Z, 2HOB, 2K7X, 2LIZ, 2OP9, 2PWX,
2Q6D, 2Q6F, 2Q6G, 2QC2, 2QCY, 2QIQ, 2V6N, 2VJ1, 2YNA, 2YNB, 2Z3C, 2Z3D, 2Z3E, 2Z94, 2Z9G, 2Z9J, 2Z9K, 2Z9L,
2ZU2, 2ZU4, 2ZU5, 3ATW, 3AVZ, 3AW0, 3AW1, 3D23, 3D62, 3E91, 3EA7, 3EA8, 3EA9, 3EAJ, 3EBN, 3F9E, 3F9F, 3F9G,
3F9H, 3FZD, 3IWM, 3J1Z, 3M3S, 3M3T, 3M3V, 3MOG, 3SN8, 3SNA, 3SNB, 3SNC, 3SND, 3SNE, 3SZN, 3TIT, 3TIU, 3TLO,
3TNS, 3TNT, 3V3M, 3VB3, 3VB4, 3VB5, 3VB6, 3VB7, 4F49, 4HI3, 4MDS, 4RSP, 4TWW, 4TWY, 4WMD, 4WME, 4WMF, 4WY3,
4XFQ, 4YLU, 4YO9, 4YOG, 4YOI, 4YOJ, 4ZRO, 4ZUH, 5B6O, 5C3N, 5C5N, 5C5O, 5EU8, 5GWY, 5GWZ, 5HYO, 5N19, 5N5O,
5NH0, 5NH0, 5NH0, 5VRF, 5WKJ, 5WKK, 5WKL, 5WKM, 5ZQG, 6FV1, 6FV2, 6JIJ
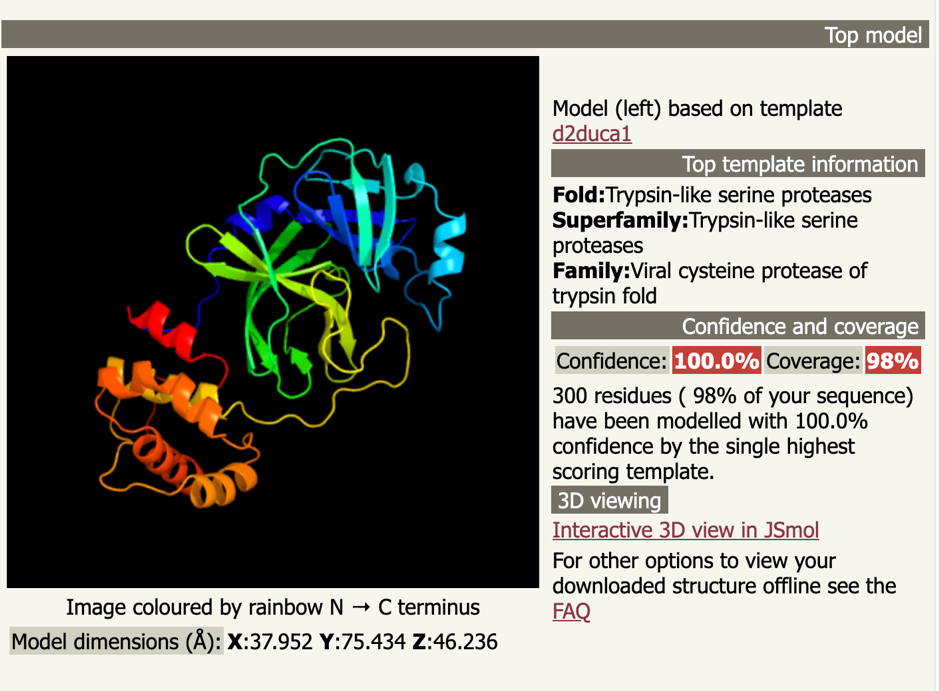
为了对2019-nCoV蛋白酶的三维结构进行建模, 我们使用了自己的Catalophore<sup>TM</sup>平台以及公共的Phyre220服务器来生成同源模型. 由于序列相似性很高, 这两种方法都获得了令人满意的结果.
Phyre2给出的最佳模型d2duca1基于折叠库中的d2duca121, d2duca1是一种胰蛋白酶样丝氨酸蛋白酶, 这种丝氨酸蛋白酶来自病毒的半胱氨酸蛋白酶, 而后者又来自SARS冠状病毒的主要蛋白酶折叠形成的胰蛋白22.

模型的Phyre2可信度为100.0%, 虽然我们不做更深入的研究.
你可以下载完整的Phyre2运行文件: d06ff0dcb8400814.tar
在后续步骤中, 我们将识别同源模型中的空腔, 并对其进行标注以生成点云.
Innophore的Catalophore<sup>TM</sup>平台以2H2Z23结构的链A为模板预测了一个同源模型, 模板来自SARS冠状病毒主蛋白酶的晶体结构, 具有可靠的N端和C端, 因此所得同源模型的综合质量较好. 我们预计这种蛋白质是一种单体. 你可以下载模型的PDB文件: <a href="https://innophore.com/wp-content/uploads/2020/01/QHD43415_1-putative-protease_cleaned.pdb_.zip">QHD43415_1-putative-protease_cleaned.pdb</a>.
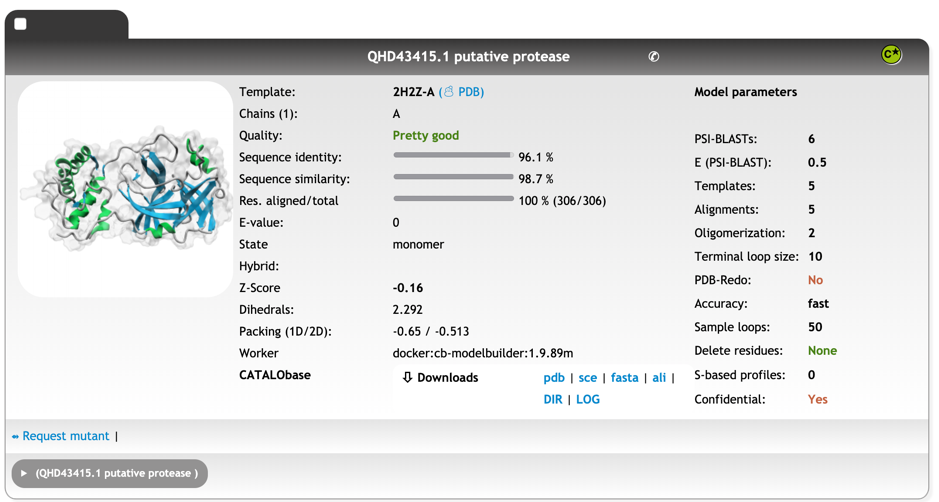
Sequence identity: 96.1 % 序列同一性
Sequence similarity: 98.7 % 序列相似性
Res. aligned/total: 306/306 残基对齐/总计
两个独立模型显示出高度相似性, 最终RMSD为0.396Å.
Match: read scoring matrix.
Match: assigning 300 x 306 pairwise scores.
MatchAlign: aligning residues (300 vs 306)...
MatchAlign: score 1616.000
ExecutiveAlign: 300 atoms aligned.
ExecutiveRMS: 7 atoms rejected during cycle 1 (RMSD=0.89).
ExecutiveRMS: 14 atoms rejected during cycle 2 (RMSD=0.62).
ExecutiveRMS: 12 atoms rejected during cycle 3 (RMSD=0.54).
ExecutiveRMS: 15 atoms rejected during cycle 4 (RMSD=0.49).
ExecutiveRMS: 9 atoms rejected during cycle 5 (RMSD=0.43).
Executive: RMSD = 0.396 (243 to 243 atoms)</pre>
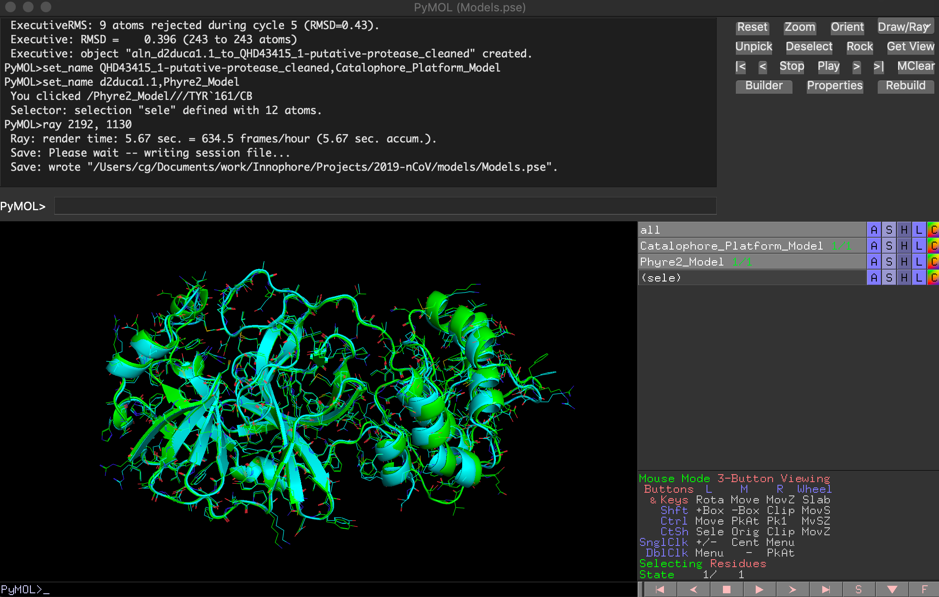
包含这两个模型的PYMOL文件可以在这里下载: 2019-nCoV_putative_protease-Models.pse
对于2H2Z模板结构, 我们的Catalophore<sup>TM</sup>数据库有6种腔体的点云腔可供使用, 这些腔是在不同环境(例如pH)下计算得到的:

对于pH为7的质子化状态, 模板有两个腔体, 均标注为EC 2.7.48:

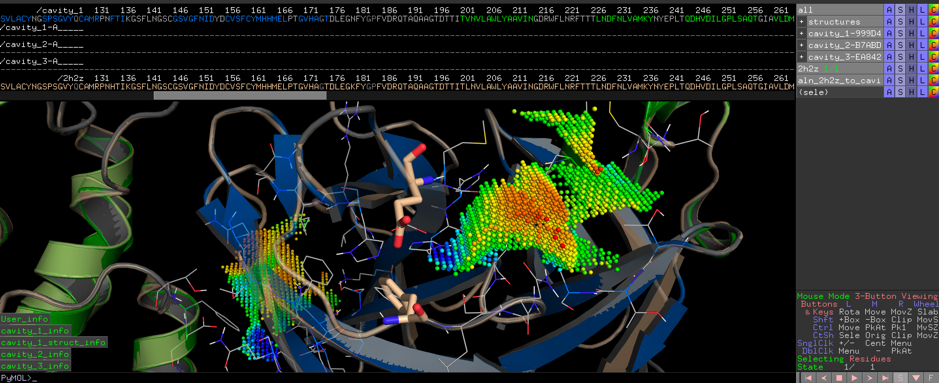
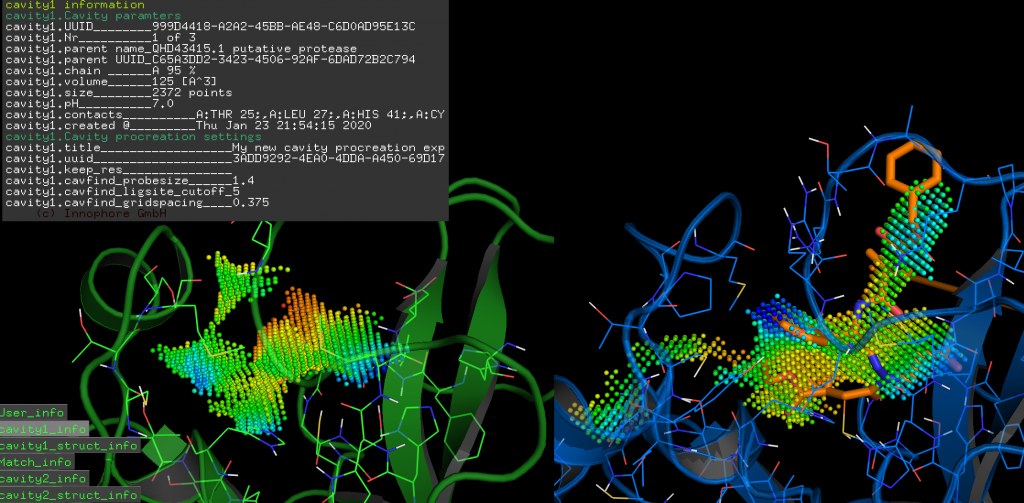
我们为推定2019-nCoV蛋白酶的可能活性位点计算了Catalophore<sup>TM</sup>点云. 使用标准设置, 我们获得了2019-nCoV蛋白酶模型的三个空腔:
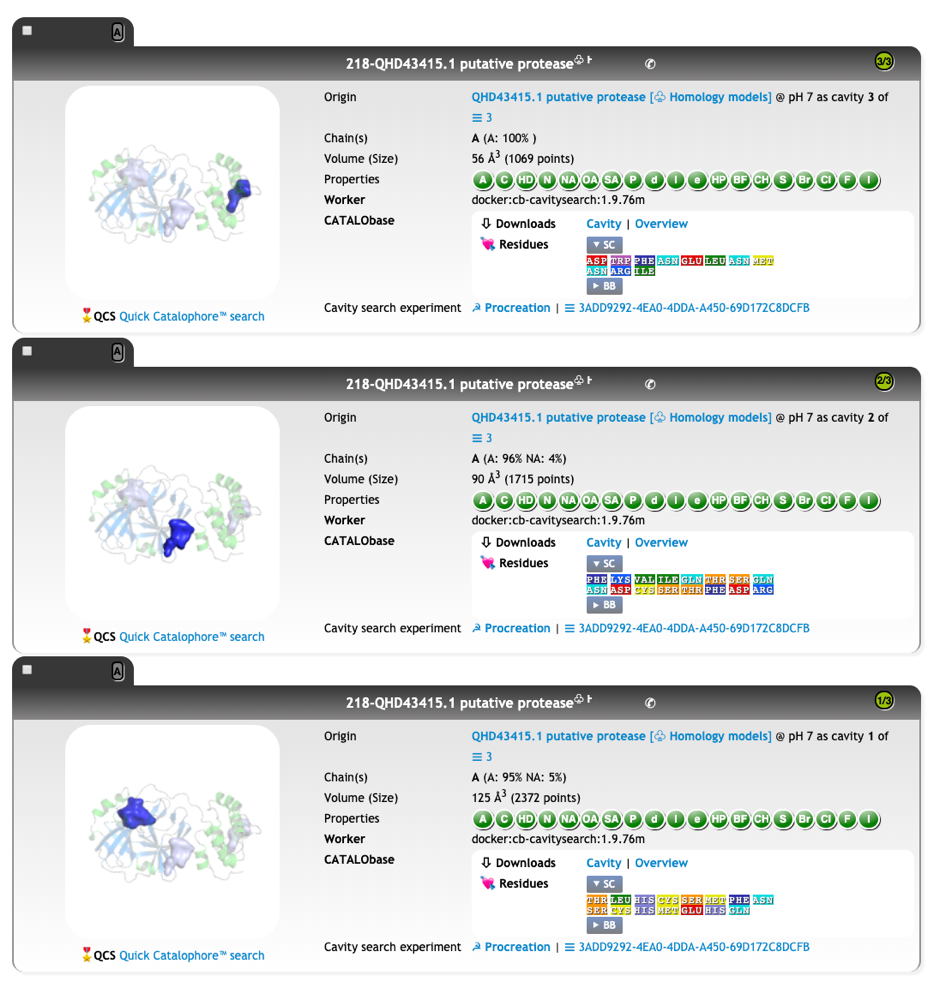
我们目前正在对最有可能的活性位点进行指纹识别, 并重新检查我们的腔体增殖参数. 我们会很快提供下载链接, 其中包括理化参数点云以及与其他冠状病毒蛋白酶的差异性分析.
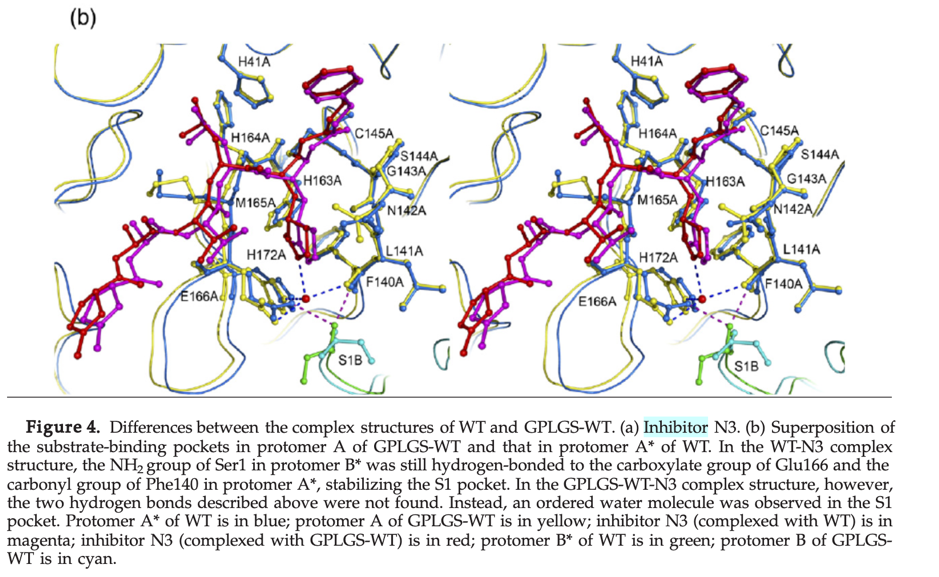
通过将模型与Yang, H.在2006年发表的PDB结构2H2Z24进行比对, 并将抑制位点的残基Glu166和Phe140(上图)映射到我们的点云Catalophores<sup>TM</sup>位点, 可以确定空腔1是一个潜在的抑制靶位点.

随后, 我们将在Catalophore<sup>TM</sup>数据库中搜索具有类似物理化学性质, 已有抑制剂的蛋白质, 从而找到可能与武汉病毒蛋白酶结合的抑制剂.

我们重新动用了3/4的计算资源(几千个核), 连夜筛选来自PDB的535879个活性腔体. 此次筛选耗费的总CPU时间约为23天.
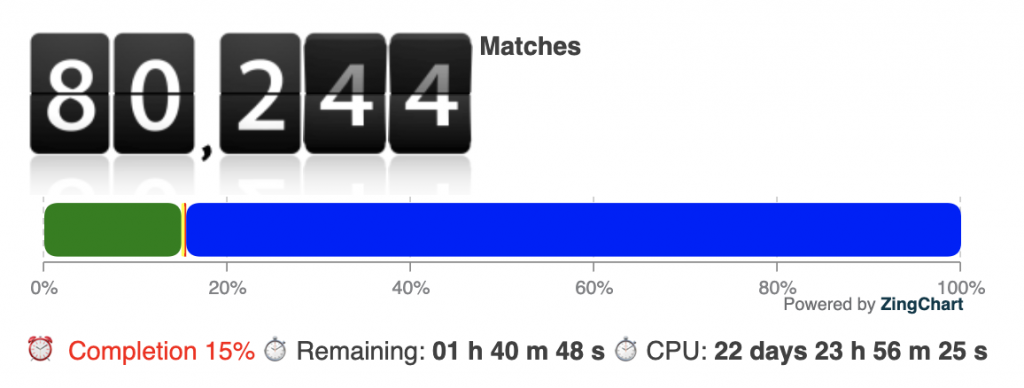
到目前为止完成了15%的筛选, 得到了几个有意义的匹配, 其中的一个腔体与来自PDB结构2A5I(Crystal structures of SARS coronavirus main peptidase inhibited by an aza-peptide epoxide in the space group C2)的点云匹配:
match-306-CDDEE285-3E3F-11EA-A0D6-0242C0D00007.zip
Matching results
Match Total score 0.03919460
Match Distance score 0.00555660
Match Overlap 1 79 %
Match Overlap 2 52 %
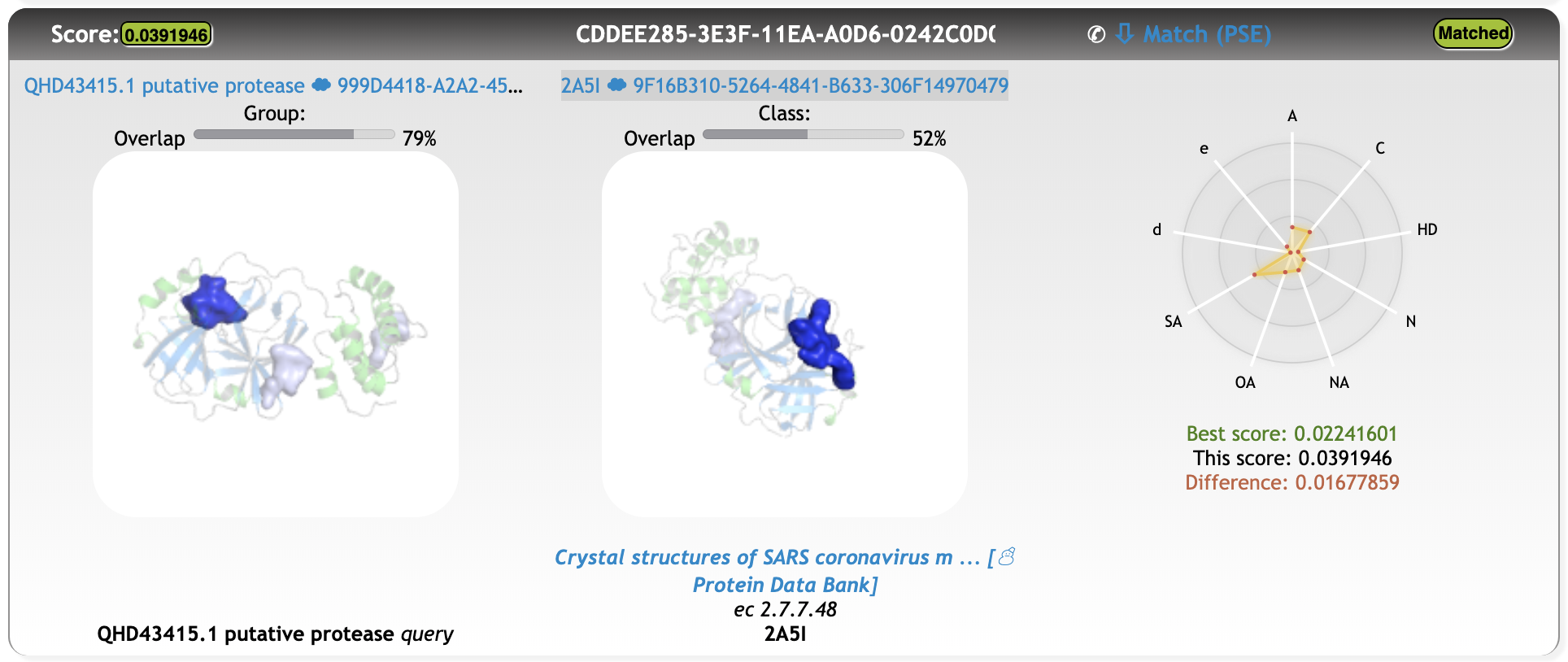
虽然腔体重叠不是很理想, 但仅仅通过腔体的旋转平移后, 蛋白质结构的对齐效果令人满意.
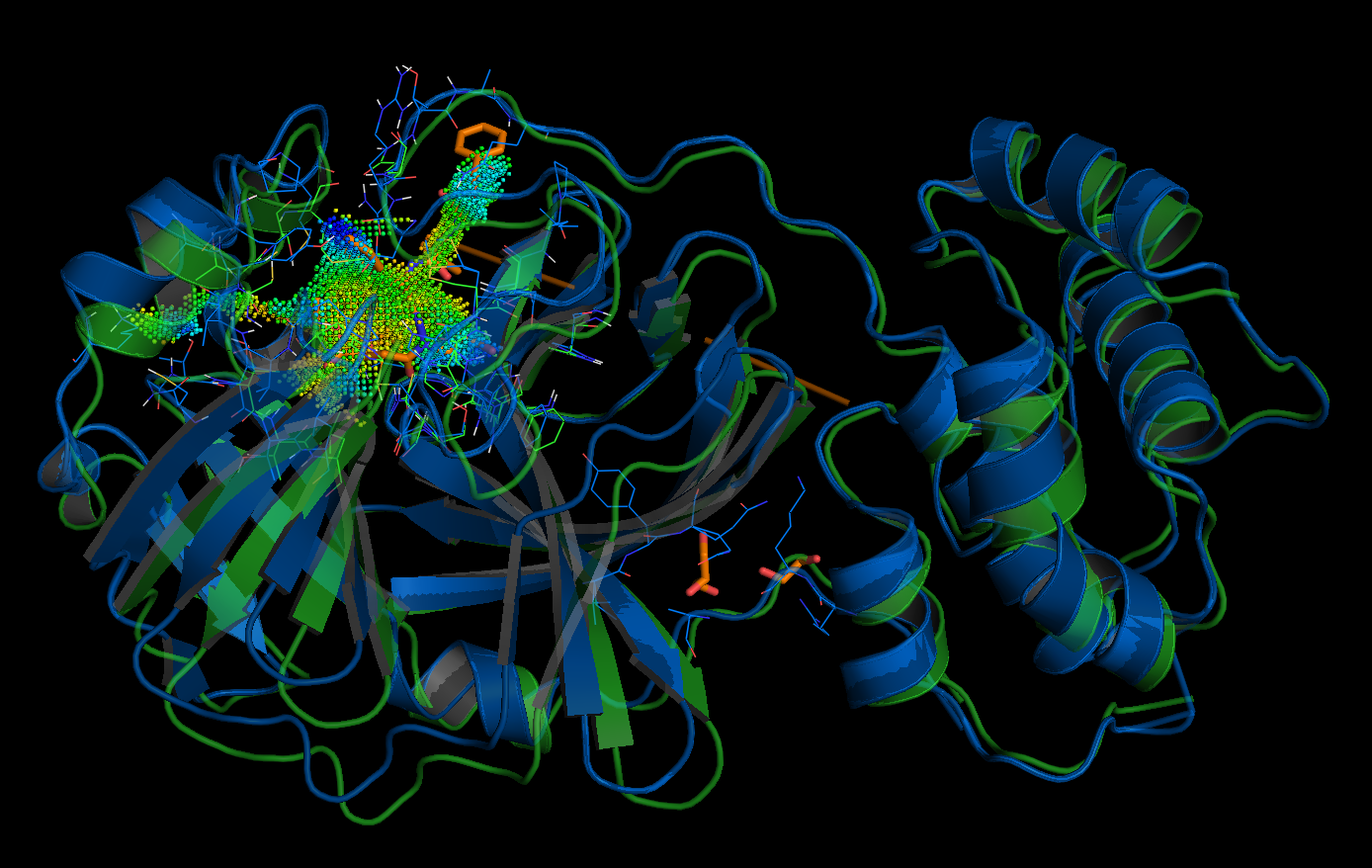
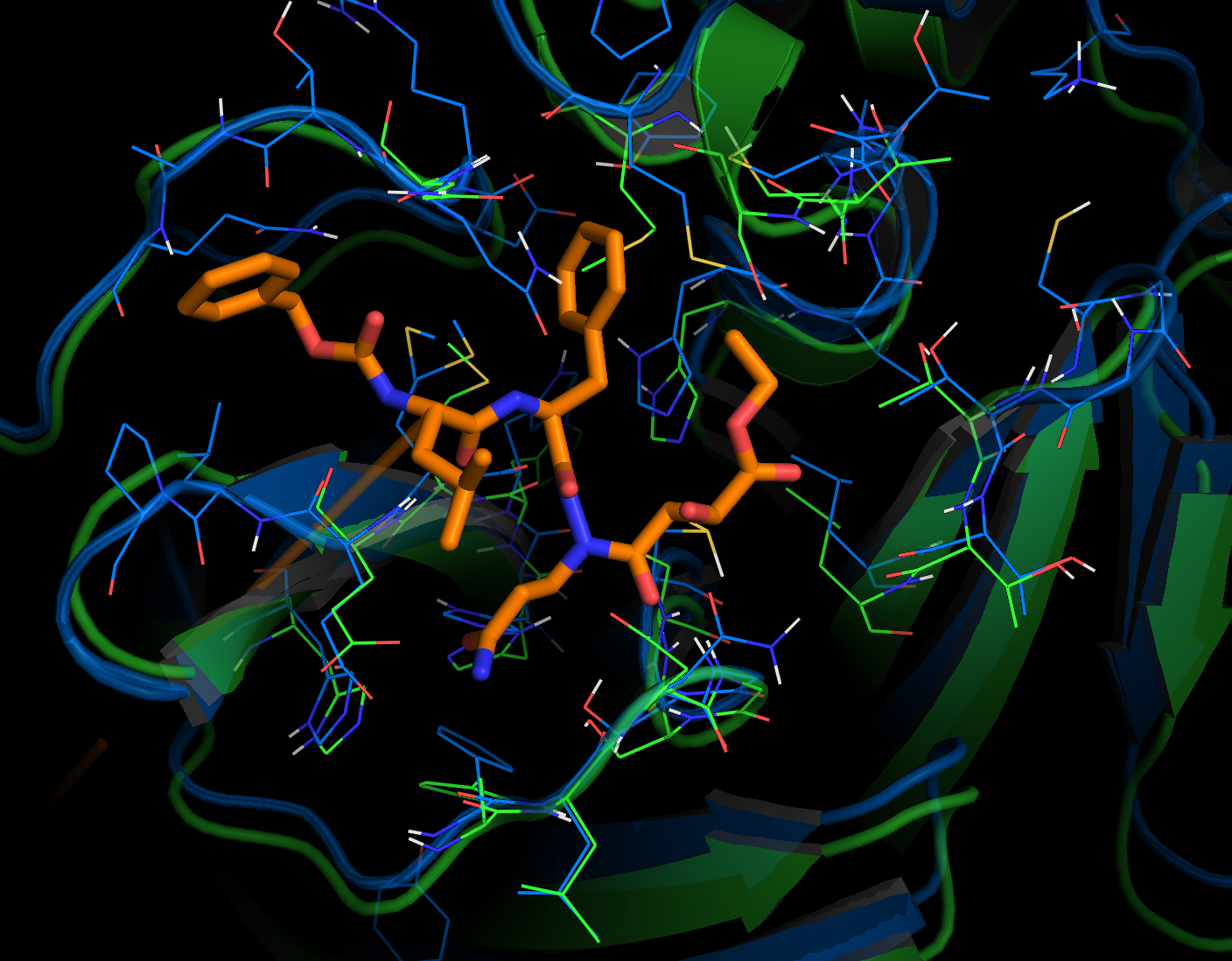
左: 假定的2019-nCoV蛋白酶的活性位点, 右: 数据库结构FDBB501F-1EF4-4AC9-8AA3-0CE76, 氮杂肽环氧化物抑制的SARS冠状病毒肽酶的点云:
腔体匹配对齐后化合物AZP的重叠. 这不是对接结果, 配体的坐标是根据活性腔体匹配进行转换后并转移到2019-nCoV蛋白酶上的.
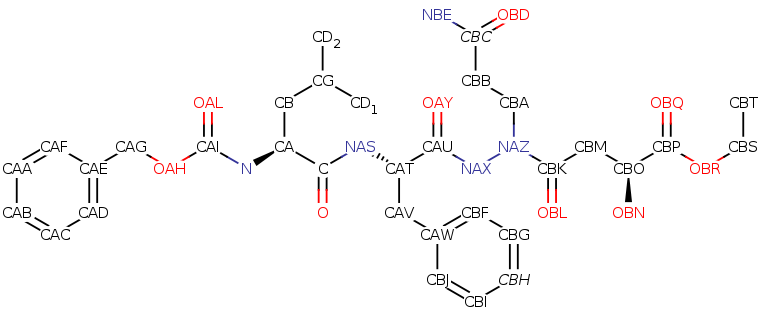
Ethyl (5S, 8S, 14S)-11-(3-amino-3-oxopropyl)-8-benzyl-14-hydroxy-5-isobutyl-3, 6, 9, 12-tetraoxo-1-phenyl-2-oxa-4, 7, 10, 11-tetraazapentadecan-15-oate
Molecular Formula: C32H43N5O9
Average mass: 641.712 Da
Monoisotopic mass: 641.306091 Da
ChemSpider: ID4450034
我们在计算机上进行的Catalophore筛选耗时1 h 38 m 21 s, 预计23 CPU days. 现在, 我们对数据库中Catalophore点云的匹配结果进行了过滤, 过滤条件是与2019-nCoV病毒蛋白酶点云有超过70%的重叠, 并且在计算腔体之前将有晶体结构的配体结合到有利的腔体中. 我们将结果限制在大于150 Å<sup>3</sup>的腔体中, 以在排名列表中更多地涵盖中到大型有机化合物类型的复杂配体.
免责声明: This following list does not take into account any pharmacological-, toxic- or side effect nor does it represent compounds directly suggested as potential drugs against 2019-nCoV. The list currently contains 148 organic compounds in total, that bind to protein cavities that share high physicochemical similarity to the 2019-nCoV protease active site cavity based on our multi-dimensional point-cloud matching.
下表列出了可能与推定的2019-nCoV冠状病毒蛋白酶结合的 前5种 化合物的初步名单, 其余候选化合物也将在进一步检查后提供:
<table id='tab-0'><caption> <input type='button' id='tab-0_tog' value='折叠表格' onclick="togtab('tab-0', this.value)"></caption><tr> <th rowspan="1" colspan="1" style="text-align:center;">Catalophore得分</th> <th rowspan="1" colspan="1" style="text-align:center;">化合物</th> <th rowspan="1" colspan="1" style="text-align:center;">分子式</th> <th rowspan="1" colspan="1" style="text-align:center;">InChIKey</th> </tr> <tr> <td rowspan="1" colspan="1" style="text-align:center;">0.023</td> <td rowspan="1" colspan="1" style="text-align:center;">G75</td> <td rowspan="1" colspan="1" style="text-align:center;"><img src='/pic/2019-nCoV/G75-270.png'></td> <td rowspan="1" colspan="1" style="text-align:center;">PIZHLOUXQJUQHF-VXNXHJTFSA-N</td> </tr> <tr> <td rowspan="1" colspan="1" style="text-align:center;">0.026</td> <td rowspan="1" colspan="1" style="text-align:center;">G82</td> <td rowspan="1" colspan="1" style="text-align:center;"><img src='/pic/2019-nCoV/G82-270.png'></td> <td rowspan="1" colspan="1" style="text-align:center;">MMMLJIROCXIHMV-XJYHXZFBSA-N</td> </tr> <tr> <td rowspan="1" colspan="1" style="text-align:center;">0.027</td> <td rowspan="1" colspan="1" style="text-align:center;">DTZ</td> <td rowspan="1" colspan="1" style="text-align:center;"><img src='/pic/2019-nCoV/DTZ-270.png'></td> <td rowspan="1" colspan="1" style="text-align:center;">KEQKAMYELZXRRN-UHFFFAOYSA-L</td> </tr> <tr> <td rowspan="1" colspan="1" style="text-align:center;">0.030</td> <td rowspan="1" colspan="1" style="text-align:center;">R30</td> <td rowspan="1" colspan="1" style="text-align:center;"><img src='/pic/2019-nCoV/R30-270.png'></td> <td rowspan="1" colspan="1" style="text-align:center;">TWIVXCFEBRGEKY-UHFFFAOYSA-N</td> </tr> <tr> <td rowspan="1" colspan="1" style="text-align:center;">0.031</td> <td rowspan="1" colspan="1" style="text-align:center;">3X5</td> <td rowspan="1" colspan="1" style="text-align:center;"><img src='/pic/2019-nCoV/3X5-270.png'></td> <td rowspan="1" colspan="1" style="text-align:center;">VZCULZJNALRGNB-DNZWLJDLSA-N</td> </tr> </table>目前, 维基百科的文章中提到了我们关于新型冠状病毒2019-nCoV的工作. 我们已经发表了2019-nCoV蛋白酶的比较模型和初步抑制剂, 我们正在与一些官方机构联系以进一步为该领域做出贡献.
感谢GISAID计划的同事, 今晚我们可以访问最近从患者那里获取的另外17个nCoV基因组数据集.
自凌晨1点以来, 我们很荣幸能与北京一家大型制药公司和中国疾病预防控制中心的生物信息学家合作, 搜索我们的Catalophore数据库以寻找可能与2019-nCoV蛋白酶结合的潜在实验药物或已批准药物, 并与我们在中国的同事一起查看我们的数据.
超过20个研究小组, 包括中国科学院上海药物研究所和上海科技大学免疫化学研究所加入了针对2019-nCoV病毒感染的应急小组, 利用积累的抗SARS药物研究经验进行了抗2019-nCoV药物研究. 他们刚刚发布了可能有效的化合物名单. 我们昨天晚上在帖子中提到的HIV-1蛋白酶抑制剂DMP450不在名单中, 但许多条目都是HIV抑制剂. CDC通知我们, Innophore昨天排名前二的候选分子G75和G82处于暂缺状态. 因此, 搜索将仅集中在已批准的药物上. 尽管如此, 通过对先前公布的晶体配合物的分析如Lee等人的研究, 这些分子仍被认为是潜在的最佳结合剂.
分子动力学分析将在4-5小时内完成.
应急小组列出的一些化合物是在配合物的晶体结构中发现的, 它们在我们昨天的Catalophore空腔搜索中排名很高, 这意味着总分低于0.1, 例如 洛匹那韦 (中国应急团队列出的前三名候选药物), 通过将2019-nCoV蛋白酶腔体与PDB结构1MUI(于2002年发布)的腔体进行匹配, 在我们的搜索中Catalophore总得分为0.085939. 因此, 洛匹那韦可能是武汉冠状病毒2019-nCoV蛋白酶腔体更好的结合剂之一, 并且是先前批准的旧药物.
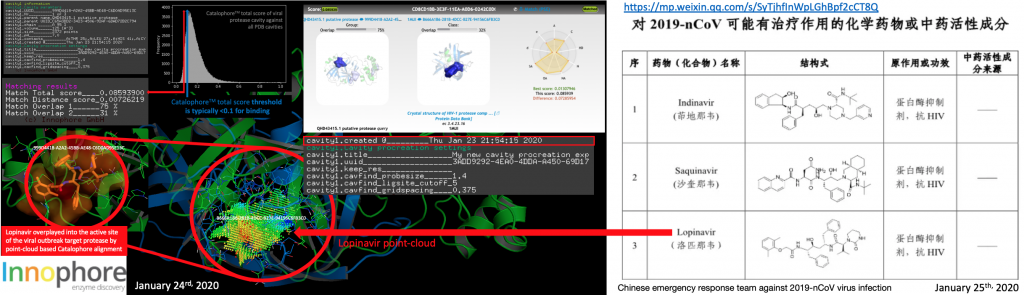
如上所述, 自凌晨1点以来, 我们正与北京一家大型制药公司和中国疾病预防控制中心的生物信息学家合作, 搜索我们的Catalophore数据库来寻找能够与2019-nCoV蛋白酶结合的潜在的实验药物或者已批准药物, 并与我们在中国的同事一起审查我们的数据. 我们必须协调文件传输以及与我们在中国的合作伙伴的沟通, 因此, 现在停止发布以完成工作. 如果你有任何建议, 请随时与我们联系, 感激不尽.
最近几天, 对我们网络服务器的要求在不断增加. 自从今晚以来, 增长速度甚至更快. 我们的网站托管给外部供应商, 如果服务器不堪负载而崩溃, 我们将迁移到亚马逊或谷歌, 并提供以下更新(如果有的话). 我们会将链接发布在LinkedIn上.
自星期六以来, 我们忙于确认, 重新评估, 筛选和交流有关ncov蛋白酶的数据. 此处严格地只引用公共媒体, 例如纽约时报昨日的报道 没有来自任何大陆的任何其他政府, 工业或科学或医疗来源.
自昨天起, 官方证实中国在医院测试洛匹那韦用于治疗冠状病毒. 洛匹那韦上周被Innophore Catalophore搜索确定为首要目标之一. 这种化合物对具有非常相似蛋白酶结构的SARS已经显示出令人振奋的结果.
我们对基于Catalopore点云比对获得的不同构象进行了(短时间)MD模拟, 并将洛匹那韦重新对接到2019-nCoV病毒蛋白酶模型中. 对接实验产生了8个可能的构象簇, 我们从其中选择了6个, 并在310 K(=36.85 ℃)下对每个构象进行了300 ps的全原子分子动力学计算. 基于对接簇, 构象被命名为LP1(最高结合能)到LP6(最低结合能). 我们稍后将发布这些轨迹.
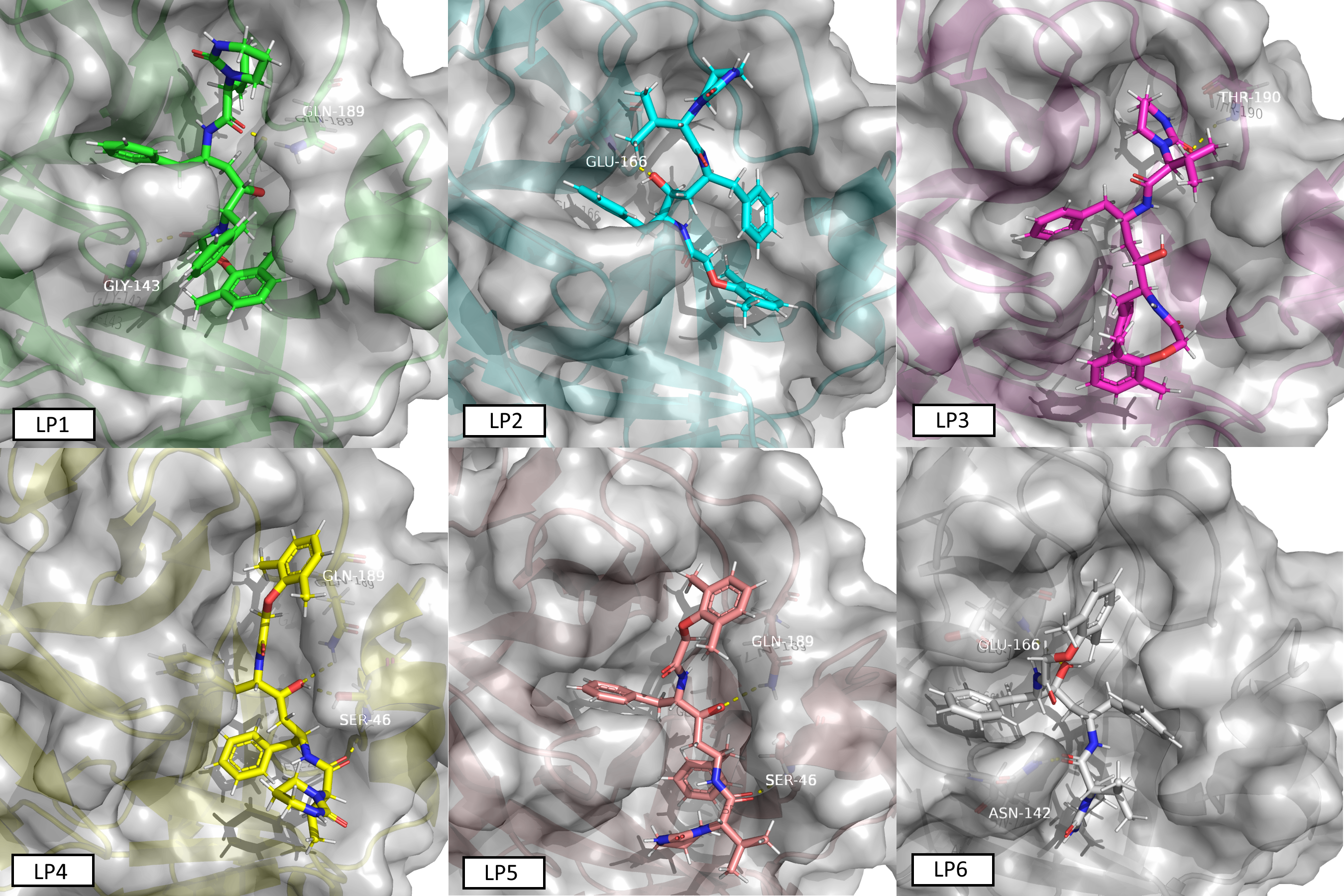 .
.
下面视频中的构象是LP1.
Xu等人发表了2019-nCoV蛋白酶模型. 他们的序列比对与我们1月23日给出的基本相同. 但是, 我们找不到要下载的结构文件. 如果任何人可以提供与我们的模型有明显差异的模型, 敬请告知.


我们为本文档创建了DOI, 你可以引用10.6084/m9.figshare.11752749; 如果你想在出版物中引用模型和渲染结果, 请引用10.6084/m9.figshare.11752752.
昨天我们开始在2019-nCoV病毒的基因组中寻找更多酶, 目前还没有任何结果.
你可以下载分子动力学模拟的所用文件.
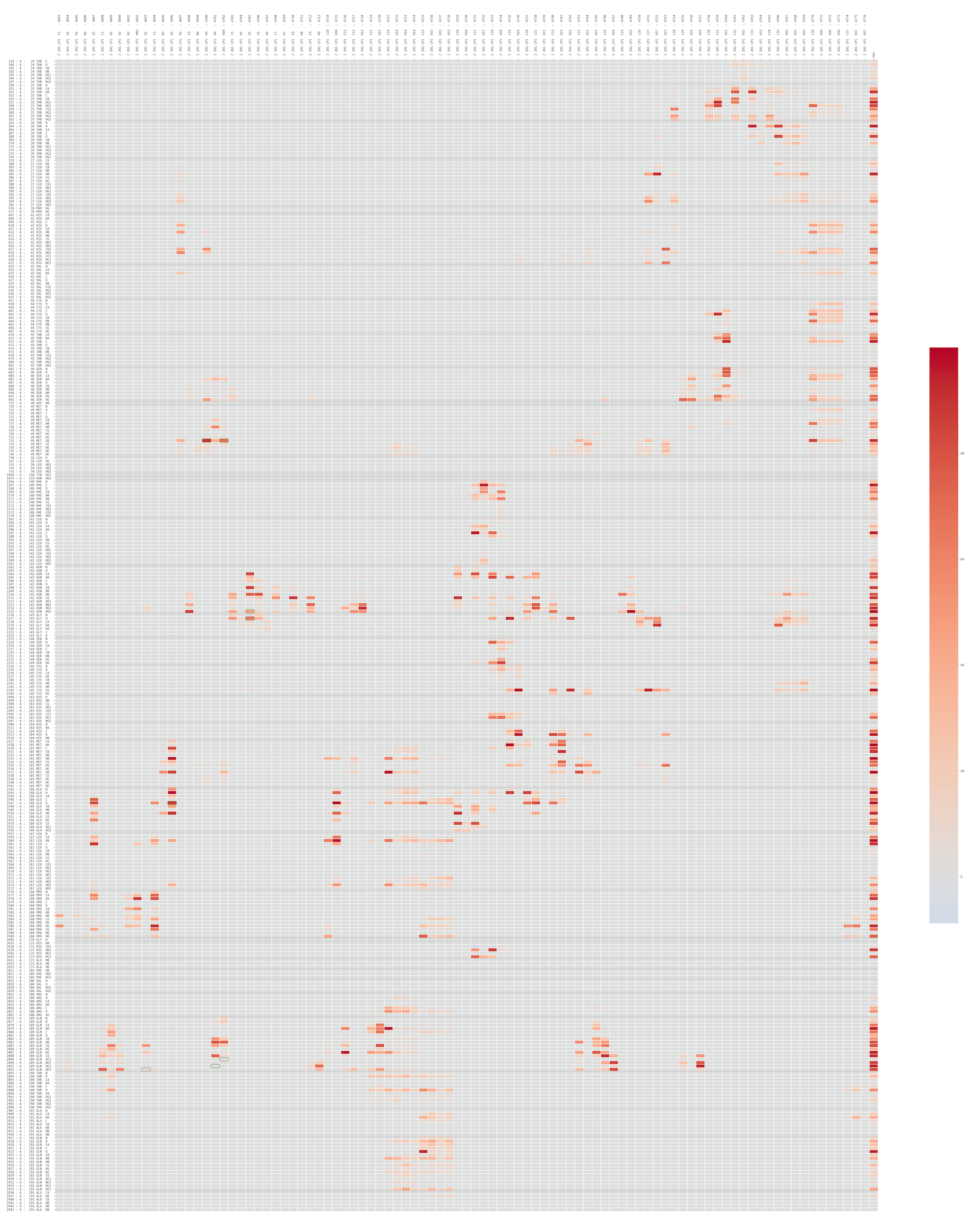
模拟所用的是2019-nCoV病毒蛋白酶Mpro与洛匹那韦的6个不同构象形成的复合物, 这是根据点云对齐并重新对接到蛋白酶模型中的. 初始的对接实验得到了8个可能的构象簇, 我们选择了其中6个在310 K(=36.85 ℃)进行了300 ps的全原子MD.
all_centroids.pse中的帧包含了对每个对接构象簇进行MD模拟后的中心.other_files目录中包含模拟产生的其他数据, 即代表配体原子与同源模型中原子的接触频率的热映射图.LP1的热映射图, 代表配体原子与同源模型中的原子的接触频率.
昨天(2020年1月31日15:45 UTC), 我们发布了一份分析(下载地址), 显示了冠状病毒2019-nCoV的蛋白酶模型与不同构象的洛匹那韦所形成的复合物在6次分子动力学模拟中的氢键网络(参见2020年1月29日16:10 UTC的更新). 结构中的虚线并不一定表示在给出的构型(模拟的平均结构)中存在氢键, 但表明该氢键在模拟期间至少存在一次. Catalophore平台生成的点云以球体模式表示, 其颜色表示嵌入度指数. 点云是根据酶的整个活性位点计算的, 没有考虑配体的存在. 氢键相互作用的列表如下. 这些数据有助于设计更好的配体. 下图显示了LP1. PyMol下载文件中包括了所有的LP.
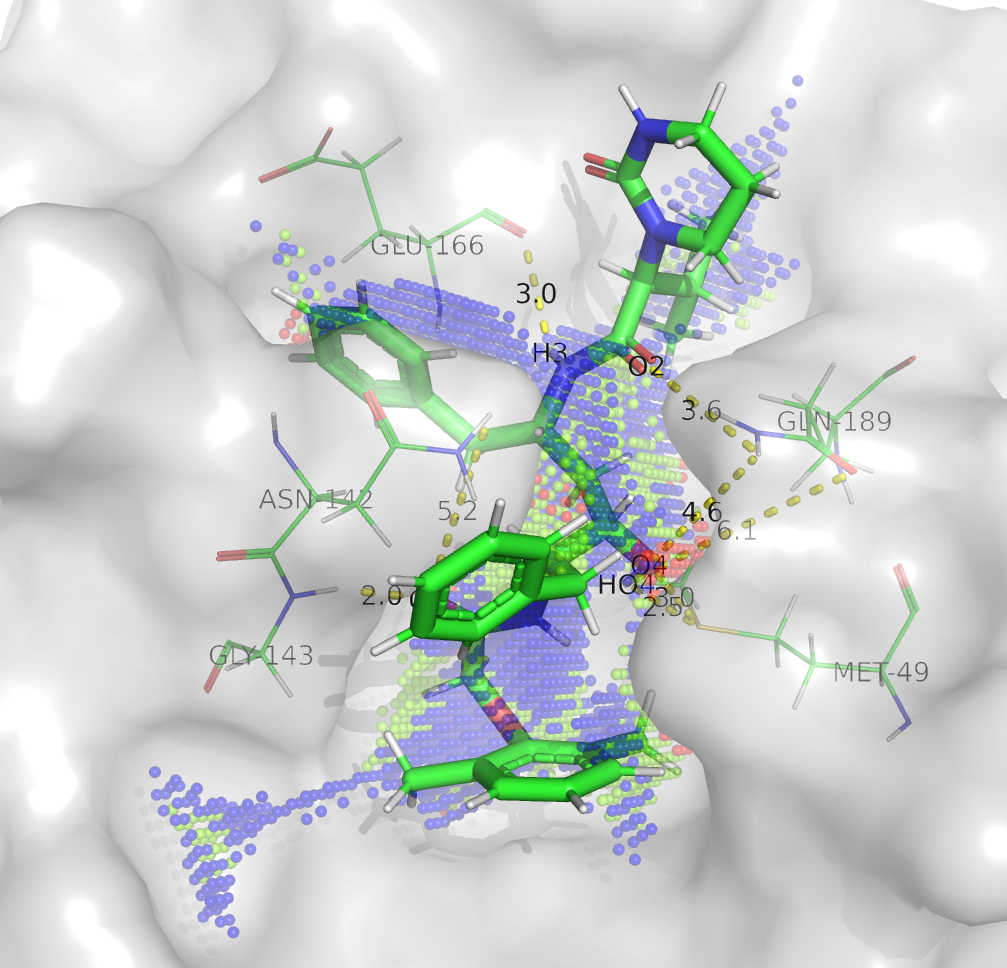
SC: 侧链, BB: 骨干
旁注: 我们仔细检查了自己的和其他组的蛋白酶对齐结果, 未确认在其他物种中发现的代表性插入.
与其他2019-ncov同源模型的比较
同时, 人们已经构建了更多的模型, 这些模型与我们模型的RMSD在0.3到0.9 Å范围内. 张课题组昨天 在其网站上发布了2019-ncov病毒所有蛋白的模型. 我们正在研究的mPro蛋白酶在其网站上称为QHD43415_5(L=306). 对齐后的RMSD为0.9 Å:
Match: read scoring matrix.
Match: assigning 306 x 306 pairwise scores.
MatchAlign: aligning residues (306 vs 306)…
MatchAlign: score 1646.000
ExecutiveAlign: 306 atoms aligned.
ExecutiveRMS: 3 atoms rejected during cycle 1 (RMSD=1.47).
ExecutiveRMS: 5 atoms rejected during cycle 2 (RMSD=1.00).
ExecutiveRMS: 4 atoms rejected during cycle 3 (RMSD=0.95).
ExecutiveRMS: 3 atoms rejected during cycle 4 (RMSD=0.92).
ExecutiveRMS: 4 atoms rejected during cycle 5 (RMSD=0.91).
Executive: RMSD = 0.886 (287 to 287 atoms)
Executive: object "aln_QHD43415_5_to_Catalophore_Platform_Model" created.
绿色: Catalophore平台, 青色: Phyre2, 黄色: I-TASSER, 粉色: SwissModel
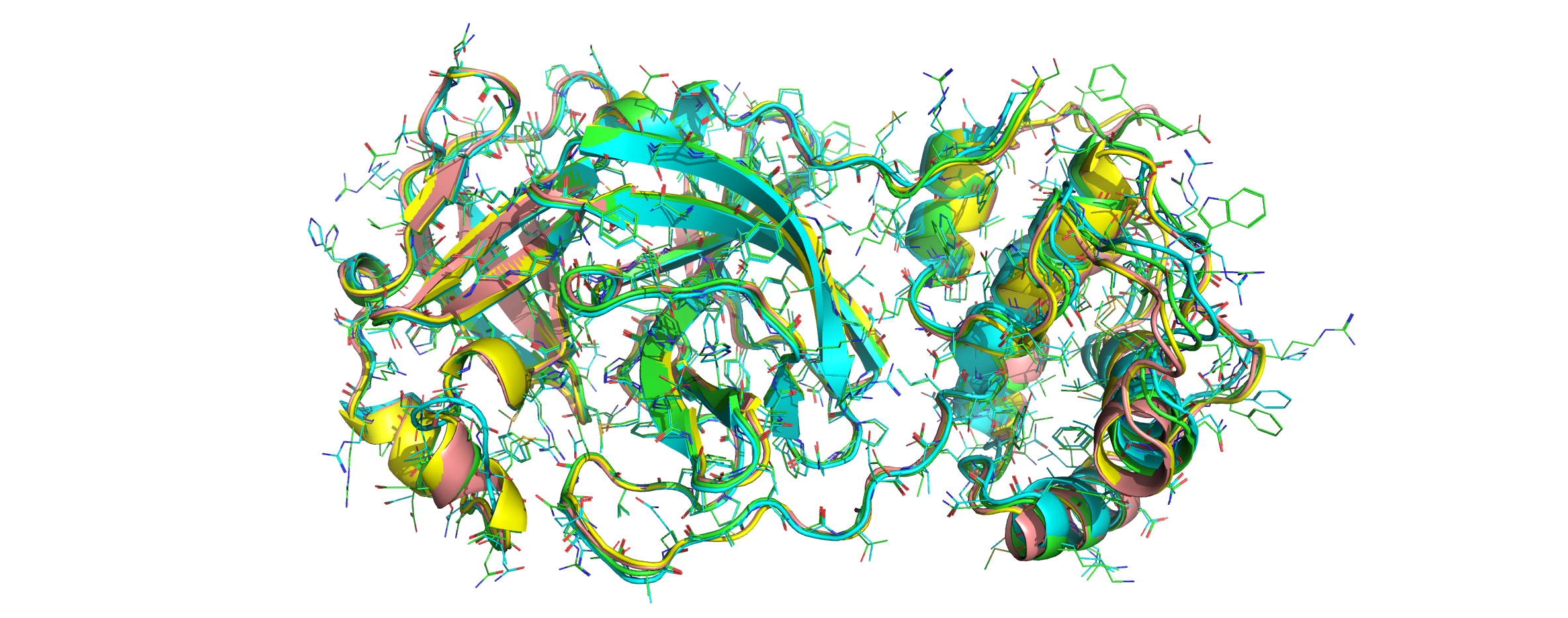
活性位点非常接近. 尽管如此, 我们将在接下来的几天中发布可用模型的详细腔体比较.
我们的合作伙伴, 来自 Universidad Católica de Murcia UCAM 结构生物信息学和高性能计算研究组 BIO-HPC 的 Horacio Pérez-Sánchez, José Pedro Cerón-Carrasco, Jorge Peña-García, Antonio Jesús Banegas-Luna 使用SBVS和LBVS方法筛选了潜在的抑制剂, 并于昨天将结果发送给我们, 以发布在我们的频道上.
A) 第一个列表(SBVS), 我们首先使用随机对接(Blind Docking)方法. 我们想要检验随机重对接计算是否能够将从蛋白质中分离出来的共结晶配体 1) 达到活性位点, 以及 2) 将其与晶体对齐后RMSD小于2 Å. 我们的BD方法已在以前的文章中进行了说明.
在BD方法的基础上, 我们同时尝试了几种对接引擎, Autodock Vina, Autodock 4, Lead Finder, FRED(Open Eyes). 我们发现FRED可以达到要求, LF也相当接近. 因此今后我们只使用这两种方法. 我们使用这两种方法筛选了DrugBank和FoodBank, 并对它们采用了一致性评分方法, 所以很短时间后, 我就可以选择这两种方法中排名前1%的化合物进行人工筛查. 然后, 我还查阅了文献并选择了表格中的那些化合物.
B) LBVS; 考虑到时间紧迫, 我采用了一种称为DBVS(Descriptor Based Virtual Screening, 基于描述符的虚拟筛选)的快速方法. 简而言之, 我们使用DRAGON计算了DrugBank, FoodBank和已知具有冠状病毒活性的待查化合物的所有描述符. 我首先使用上海发布的晶体结构针对这两个数据库进行了筛选, 只有具有完全相同值的匹配的描述符之间的欧几里得距离小于10的情况下, 我才会选择它们进行进一步的人工筛查并查阅文献.
在这个LBVS方案中, 我们还会应用下下面的方案Optimizing Electrostatic Similarity for Virtual Screening: A New Methodology
免责声明: This following list does not take into account any pharmacological-, toxic- or side effect tested in clinical trials nor does it represent compounds directly suggested as potential drugs against 2019-nCoV. Innophore is not taking responsibility (please see our disclaimer on the top of the page).
我们昨天收到的列表可以从此处下载.
我们的合作伙伴, 来自上海科技大学的Yang课题组正式发布了蛋白酶的晶体结构. 下图给出了对齐的结构, 我们也更新了包含晶体结构的DOI数据集
黄色: X射线, 其他颜色: 模型
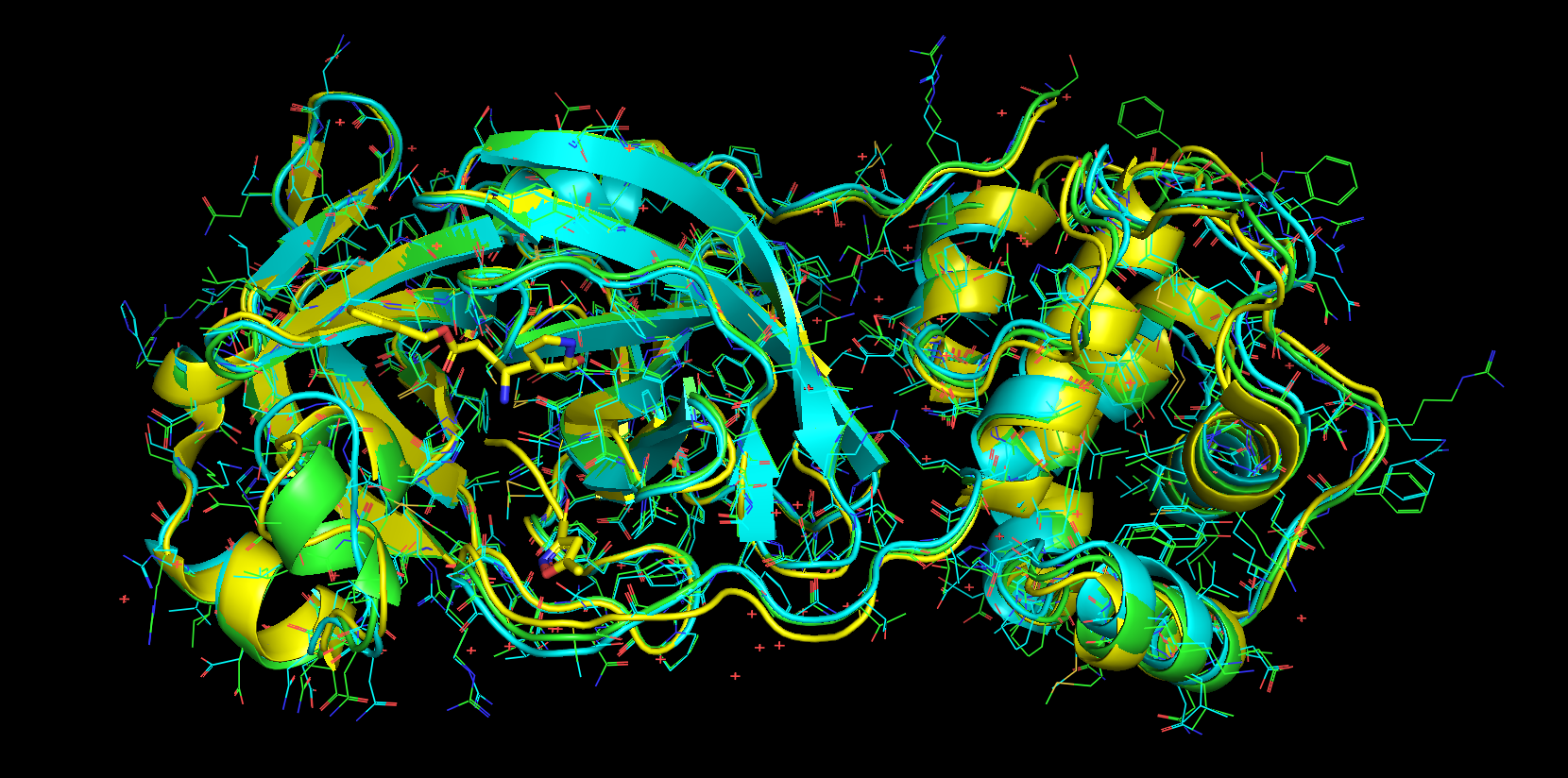
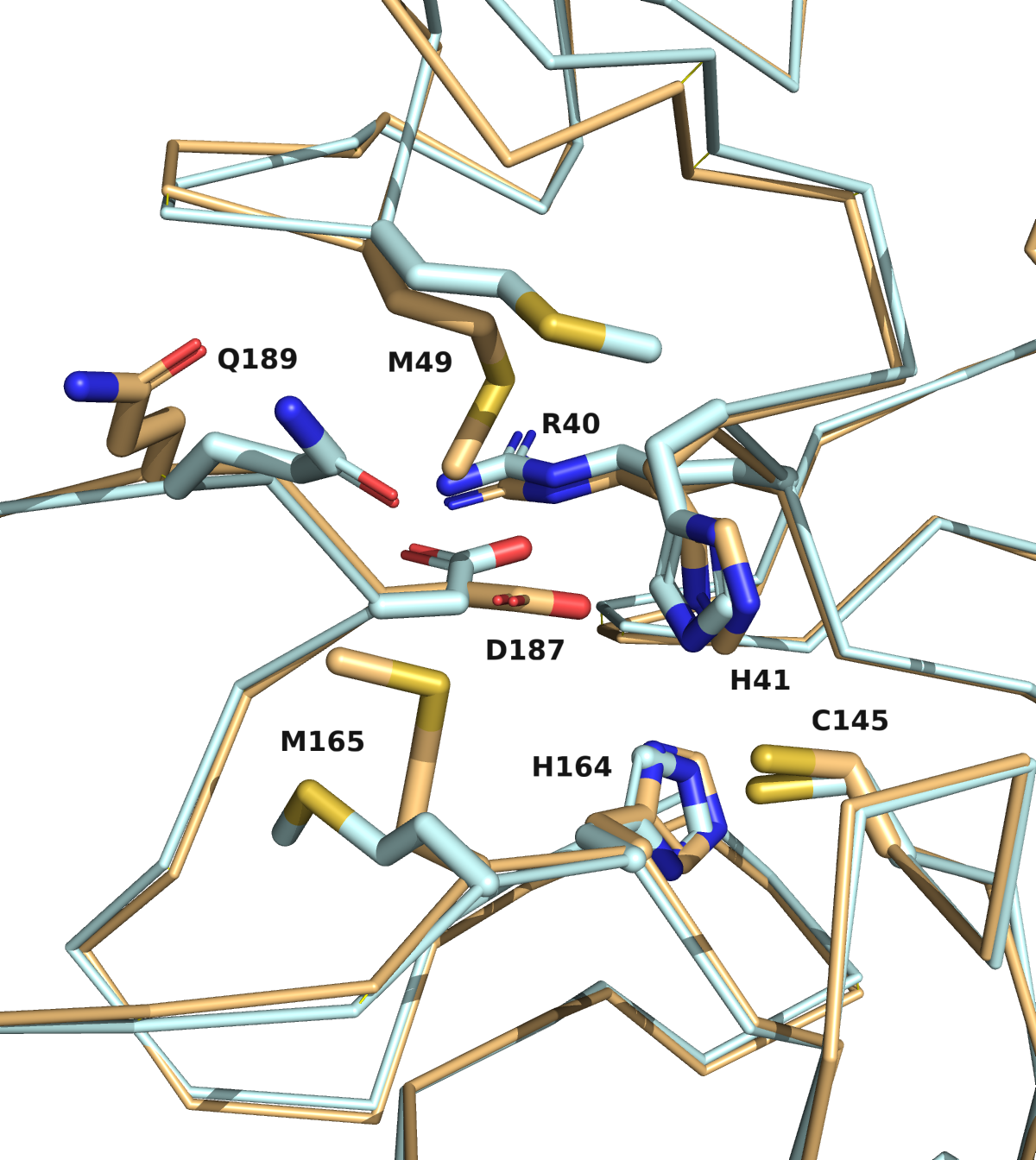
我们将很快发布过去8天中我们用晶体结构得到的MD, catalophore和对接数据. 总的来说, nCov蛋白酶现有的晶体结构与之前Innophore给出的同源模型非常相似. 对306个叠合很好的的Cα原子中的282个, 对齐后的RMSD为0.6埃. 正如预期的那样, 一些侧链, 特别是蛋白质表面的侧链, 在这两种结构中的构象不同. 这种差异也存在于推定活性位点内的残基中(见下图: 橙色为Innophore模型, 青色为X射线结构). 特别地, 如果未考虑的话, H41和D187的不同构象可能会影响到对接计算.
我们必须齐心协力. 特殊情况需要特殊的努力. 我们非常荣幸能够协调和管理国际合作, 并在University of Graz的帮助下分配大量资金, 以对抗目前的2019-nCoV爆发. 综合模拟技术和体外技术进行系统研究, 在以新技术为基础的最先进的既定方法之间进行仔细平衡, 我们旨在快速发现和测试抑制2019-nCoV M(pro)蛋白酶的化合物, 这是我们的主要目标. 在欧洲主要HPC设施的支持下, 活跃于生物信息学, 药物筛选和设计领域的全球领先机构将与高通量的体外和体内实验验证方法相互验证. 该联盟涵盖了整个流程, 从患者分离的2019-ncov基因组到在2019-ncov细胞培养物中进行化合物测试. 我们的网站(目前仅列出合作伙伴)昨天上线fastcure.net
昨天, 我们以公开数据发布了SARS-CoV-2(以前为2019-nCoV)主要蛋白酶(Mpro)的一系列分子动力学模拟. 我们邀请你使用这些数据进行进一步分析.
我们的合作伙伴, 上海科技大学的Yang教授在310 K下对SARS-CoV-2 Mpro晶体结构进行了3次长度为0.5 ns的分子动力学模拟, 模拟中Mpro与不同构型的潜在配体形成复合物. 我们现在可以使用大量的计算资源, 并将继续进行其生物信息学分析.
https://www.who.int/publications-detail/surveillance-case-definitions-for-human-infection-with-novel-coronavirus-(ncov)↩
https://edition.cnn.com/2020/01/23/china/wuhan-coronavirus-update-intl-hnk/index.html↩
https://www.cbc.ca/news/health/coronavirus-human-to-human-1.5433187↩
https://www.reuters.com/article/us-china-health-pneumonia-who-idUSKBN1ZD16J↩
https://en.wikipedia.org/wiki/Novel_coronavirus_(2019-nCoV)↩
https://link.springer.com/protocol/10.1007%2F978-1-4939-2438-7_1↩
https://www.asiatimes.com/2020/01/article/wuhan-disease-spread-by-bats-animals-expert/↩
https://www.webmd.com/lung/news/20200122/what-to-know-about-new-coronavirus-from-china↩
https://bmcgenomics.biomedcentral.com/articles/10.1186/1471-2164-15-S7-S5↩
https://www.biorxiv.org/content/10.1101/2020.01.20.913368v1↩
https://www.biorxiv.org/content/10.1101/2020.01.20.913368v1↩
https://www.ncbi.nlm.nih.gov/nuccore/MN908947.3?location=266:13468,13468:21555↩