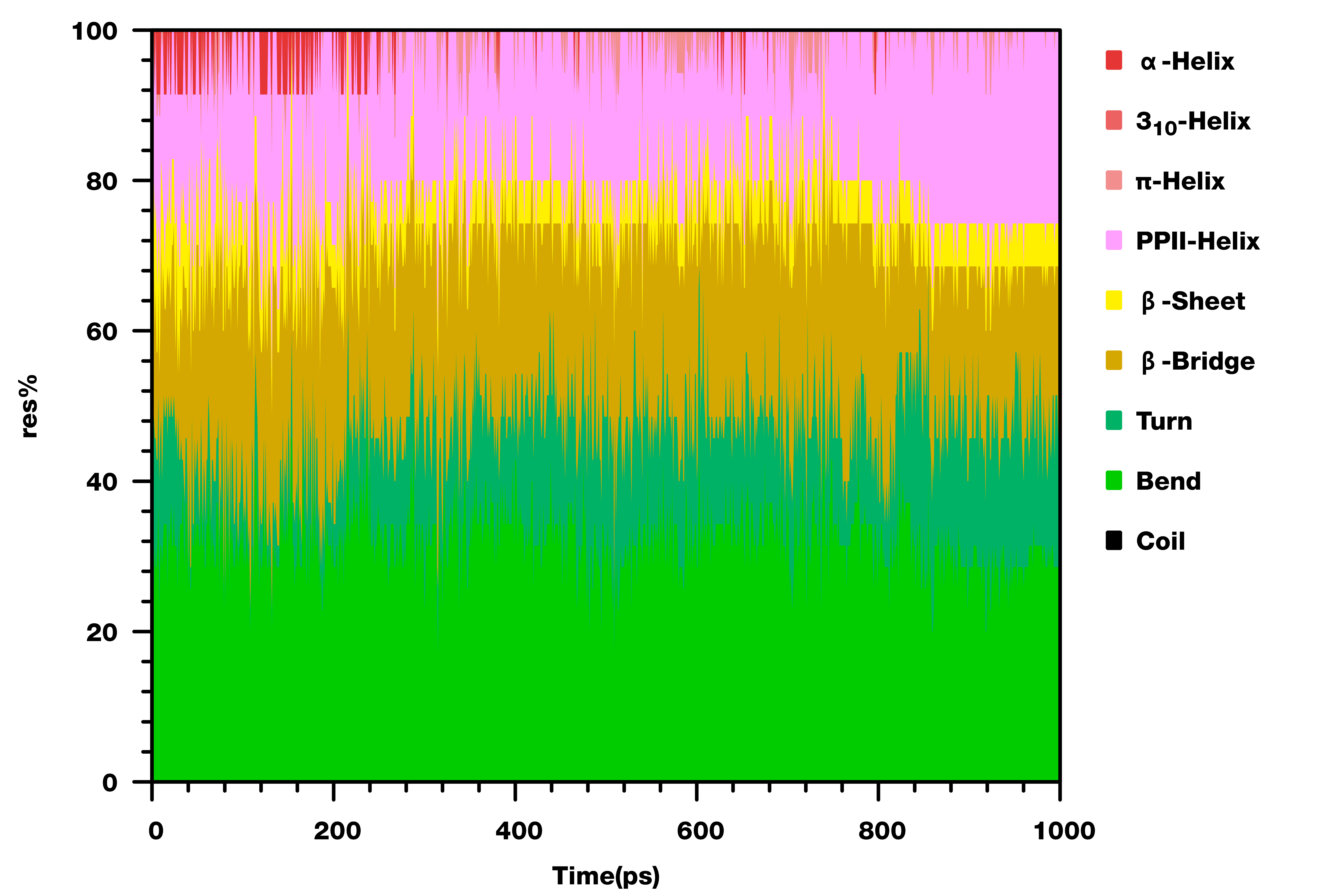
蛋白二级结构随时间的演化可用于表征蛋白折叠是否稳定. GROMACS 2023用于计算蛋白二级结构的工具为dssp. 这个工具内置了计算二级结构的代码, 无须再借助外部工具, 并且使用了新版本DSSP算法, 可以识别PPII螺旋. 为此, GROMACS 2019中的do_dssp工具已经废弃.
由于这个工具是新增的, 功能还不完善, 有些do_dssp具有的功能尚未实现. 工具目前只能给出二级结构的数据, 而无法直接给出以前的xpm图. 所以我们需要借助其他程序或脚本来绘制二级结构演化图.
我以前写过一个脚本xpm2all, 可以将xpm二级结构数据转换为gnuplot绘图脚本, 但它没有办法直接处理dssp的输出数据. 为此, 我修改了一下原脚本, 使支持直接读取dssp所给的数据文件, 并转换为gnuplot绘图脚本. 此外, 还更新了一下绘制模式, 加了几种颜色方案. 由于改动较大, 我将其从xpm2all中抽离出来, 成为单独的一个工具, 称为dssp2gp, 与xpm2all并列放在一起.
>>>>>>>>>>>>>>>> dssp2gp <<<<<<<<<<<<<<<<
>>>>>>>>>>>>>>>> Jicun Li <<<<<<<<<<<<<<<<
>>>>>>>>>> 2024-01-11 23:12:11 <<<<<<<<<
Usage: dssp2gp <file.dat|file.xpm> [-DRAW] [-COLOR] [RANGE]
[-t0 t0] [-dt dt] [-tu tu]
Default: dssp2gp dssp.dat -xyz -pdb 1:1E9:1 -t0 0 -dt 1 -tu ps
option: DRAW:
-xyz: xyz data
-box: plain box
-fancy: fancy helix and beta sheet
COLOR: gmx, vmd, pdb, rcsb, taylor, p1, p2, p3, p4
RANGE: [minRes:maxRes:ystart]
minRes: start from #residue
maxRes: end with #residue
ystart: y value of minRes
-t0: starting time
-dt: timestep
-tu: unit of time, fs, ps, ns
--------------------------------------------------------------------------------"
dssp的时间选项类似.-xyz, -box, -fancy, 颜色方案选项-gmx, -vmd等.起始残基:终止残基:起始编号涉及残基编号在各个工具间的转换, 较麻烦, 要考虑多种情况. dssp输出的数据没有残基编号信息, 可以认为是1:残基数. 起始残基:终止残基的指定基于dssp的编号系统, 因而其可取范围为1:残基数. 但计算所用蛋白中的残基编号可能不是从1开始的, 原因可能在于计算时只选择了整个蛋白的一部分, 或者原始蛋白PDB中的编号就不是从1开始的. 如果存在这种不一致, 那就可以设置绘制时的起始编号.设想有个蛋白共100残基:
1:100, pdb2gmx时默认会使用原始编号, 则所得拓扑文件中的残基编号同样为1:100``.dssp分析蛋白时重新编号为1:100,dssp2gp`转换时选择所有残基且使用原始编号绘图, 所有编号都一致, 所以无须设置范围选项.若蛋白晶体PDB中原始编号13:112, pdb2gmx时未重新编号, dssp分析蛋白时只选取了其中的30个残基, 其原始编号为20:49. dssp分析时将其重新编号1:30, dssp2gp转换时只使用其中的10个残基, 11:20, 但绘制时希望使用原始PDB中的编号, 则范围选项应设为11:20:30.
#: 1 2 ... 8 9 ... 18 19 ... 26 27 ... 36 37 ... 99 100
PDB: 13 14 ... [20 21 ... 30 31 ... 38 39 ... 48 49] ... 111 112
pdb2gmx: 13 14 ... [20 21 ... 30 31 ... 38 39 ... 48 49] ... 111 112
dssp: [ 1 2 ... 11 12 ... 19 20 ... 29 30]
dssp2gp: [11 12 ... 19 20]
几种不同的颜色方案的对比

-fancy绘制模式只适用于少量关键蛋白的二级结构分析, 因为数目过多时无法区分.
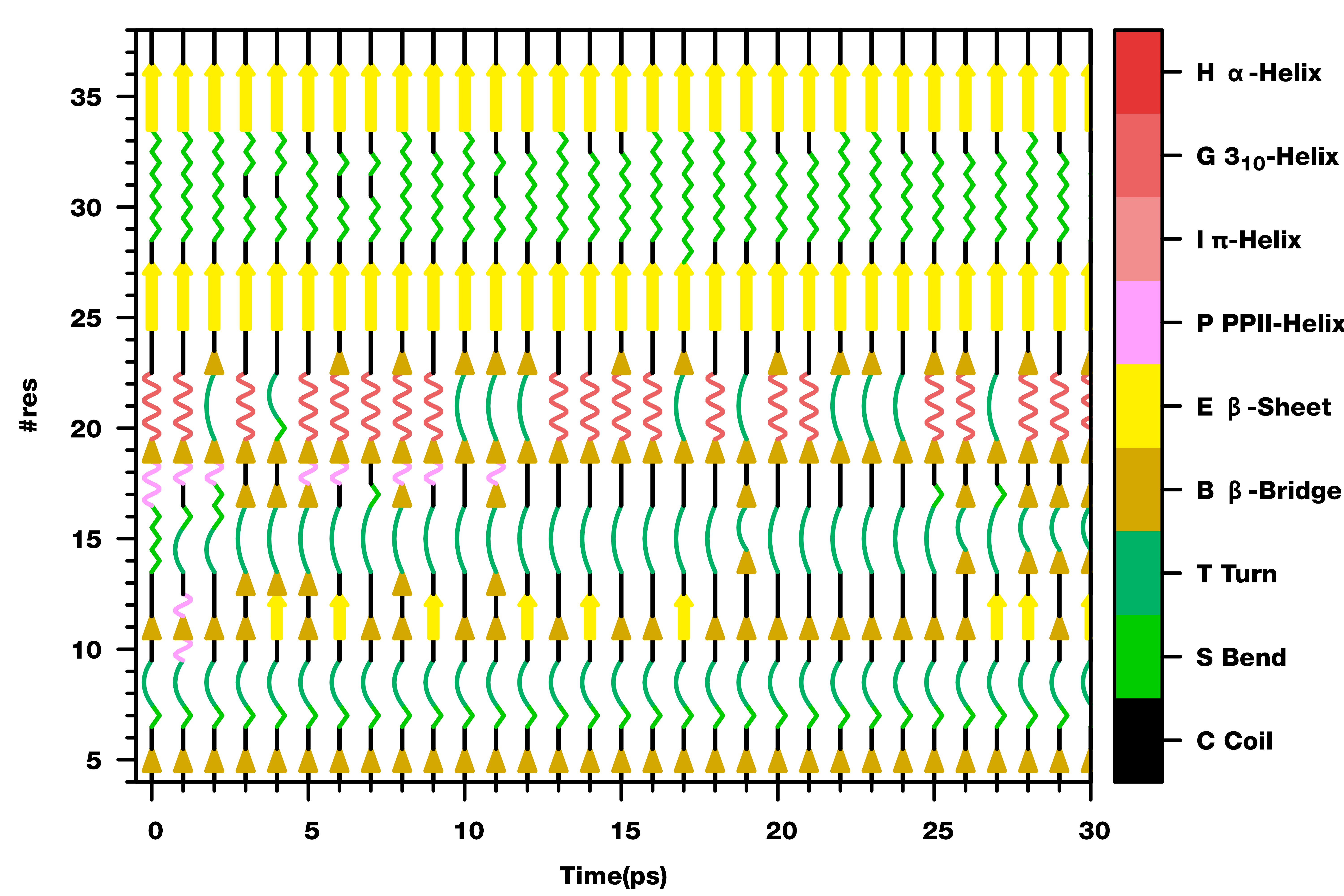
二级结构含量